Investigación uruguaya identifica nuevas variantes genéticas asociadas al síndrome de Lynch
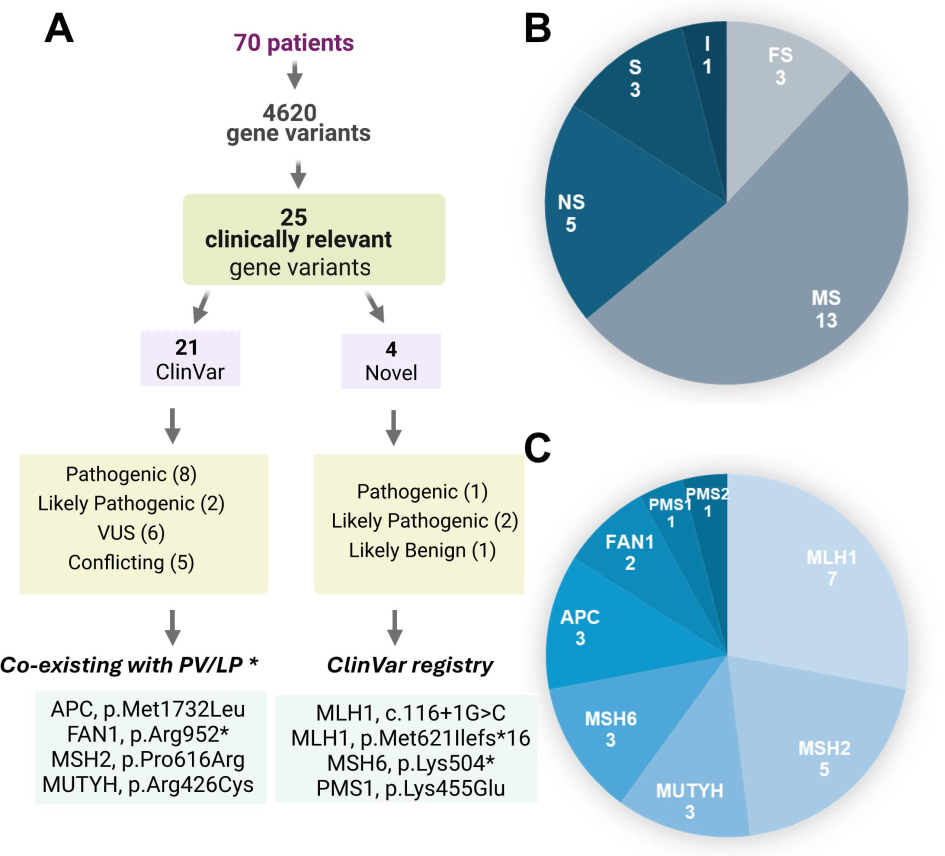
El artículo, recientemente aceptado para publicación en Frontiers in Oncology, caracterizó genéticamente a setenta pacientes no relacionados, utilizando una plataforma de secuenciación masiva de última generación (NGS, por sus siglas en inglés) y un panel de genes diseñado especialmente para este estudio. La investigación permitió identificar variantes genéticas patogénicas o potencialmente patogénicas en el 35% de los casos, incluyendo cuatro variantes nuevas no reportadas previamente en bases de datos internacionales.
El síndrome de Lynch es responsable de entre el 1 y el 7% de todos los casos de cáncer colorrectal y está causado por mutaciones en genes del sistema de reparación del ADN. La detección temprana de estas variantes es fundamental para la orientación genética y la prevención en familiares en riesgo. En este trabajo, se combinaron técnicas moleculares de alta sensibilidad con análisis clínicos y genealógicos, destacando el potencial de los paneles genéticos personalizados para la atención oncológica en sistemas de salud de mediana escala.
El estudio fue desarrollado por investigadoras e investigadores del Departamento de Genómica del IIBCE, de la Facultad de Medicina (Unidad Académica de Genética de la Facultad de Medicina, y el Dpto. Básico de Medicina del Hospital de Clínicas) y la Facultad de Ciencias de la Udelar. Participaron activamente médicos del Centro de Oncogenética Uruguayo del Banco de Tumores del Hospital Central de las Fuerzas Armadas. Todo el estudio se realizó en Uruguay, el ADN de los pacientes fue secuenciado en la Plataforma de Secuenciación Masiva del IIBCE.
Además de validar variantes conocidas, el equipo identificó y reportó nuevas variantes en los genes MLH1, MSH2, MSH6, MUTYH y PMS1, contribuyendo así al conocimiento global sobre las bases genéticas del síndrome en poblaciones latinoamericanas.
Este trabajo destaca la importancia de desarrollar capacidades locales en genómica para fortalecer la medicina personalizada y la prevención del cáncer en Uruguay.
___________________________________________________________________
La imagen se extrajo de la publicación del artículo.
Corresponde a las variantes clínicamente relevantes identificadas.
(A) Resumen de los resultados de la clasificación de variantes.
*Variantes genéticas que coexisten con PV/LP (patógenas/probablemente variantes patogénicas) en un gen diferente.
(B) Distribución de tipos de mutación en MS (error), NS (sin sentido), FS (cambio de marco), S (empalme) e I (intrónico).
(C) Distribución de mutaciones en los genes analizados.
